Chapitre II
La RMN HRMAS et la synthèse organique en phase solide
1 Introduction
1.1 Les techniques danalyses utilisées en phase solide
Un des premiers freins au développement de la synthèse en phase solide est le manque de techniques danalyses rapides et précises lorsque la molécule est encore ancrée sur le support solide. La méthode la plus utilisée pour la caractérisation des molécules synthétisées en phase solide consiste à séparer la molécule de son support solide pour procéder à la purification de la molécule et ensuite caractériser celle-ci par les méthodes danalyses classiques telles que la CCM, la RMN liquide haute résolution ou la spectrométrie de masse. Cette méthode est très efficace mais présente deux inconvénients, dune part elle est destructrice pour le support qui ne peut pas être réutilisé et dautre part, létape de purification nécessite beaucoup de temps. De plus la coupure chimique peut altérer la molécule synthétisée, et/ou les intermédiaires de synthèse peuvent présenter des problèmes de solubilité ou dinstabilité.
Néanmoins, lorsque le composé est encore ancré sur le support, il existe certaines méthodes qualitatives de contrôle basées sur des tests colorimétriques. Lors de la synthèse peptidique par exemple, les étapes de couplage ou de déprotections peuvent être contrôlées notamment par le test à la ninhydrine[1],[2]. Lors de tels tests, on peut sinterroger sur leur sensibilité : jusqu'à quel niveau ces techniques sont applicables. De plus, avec ces tests, on obtient un résultat global, sans aucune information au niveau moléculaire. Si lon considère le cas dun acétylation irréversible dune fonction amine lors de la synthèse dun peptide. Le test ninhydrine après létape de couplage donnera un résultat négatif, ce qui confirme la disparition de lamine primaire, mais en aucun cas on ne peut savoir que lacide aminé ancré ne sera plus réactif pour les réactions suivantes. Le problème des tests colorimétriques et quils ne sont pas généraux, ils permettent seulement de mettre en évidence quelques fonctionnalités caractéristiques.
Avant de choisir une technique danalyse, lexpérimentateur doit sinterroger sur le but de lanalyse, la quantité de produit nécessaire et aussi la possibilité de réutilisation du support. Nous allons maintenant présenter différentes méthodes danalyses de molécules ancrées sur un support solide avec leurs avantages et leurs inconvénients.
Différentes méthodes basées sur la spectrométrie de masse permettent une analyse de molécules ancrées sur le support solide. La spectroscopie MALDI-TOF (Matrix-Assisted Laser Desorption Ionization Time Of Flight) consiste à séparer à laide dun laser, la molécule synthétisée du support solide. [3] Cette technique nécessite lutilisation dune résine particulière car lancrage doit être photosensible. Après libération du composé au sein du spectromètre il est analysé classiquement par spectrométrie de masse. Lavantage de cette méthode est quelle est très sensible et ne nécessite que très peu de matériel, quelques billes de résine suffisent. Par contre, le matériel utilisé est perdu pour la suite de la synthèse. Cette technique permet une identification des molécules mais ne permet pas de quantifier les différentes espèces. La spectrométrie de masse de type SIMS (Secondary Ion Mass Spectroscopy) est une technique qui permet lanalyse de la molécule ancrée tout en conservant lintégrité du support. Récemment Aubagnac et al[4] ont développé une méthode danalyse non destructrice faisant appel à cette technique. Elle est particulièrement utile pour les nouveaux supports solides tels que les PINS et ne nécessite pas lemploi de « linker » photosensible. De plus cette méthode danalyse permet didentifier les molécules mais aussi une quantification directe sur le support[5]. Cette méthode consiste à bombarder la cible par un faisceau dions, puis le temps de vol des ions incidents (ions secondaires) est mesuré. Ces ions sont caractéristiques de la surface de léchantillon étudié.
LInfra Rouge à Transformée de Fourrier (IR FT) permet dévaluer directement sur une résine le taux de déprotection lors de la synthèse peptidique à condition que les groupes protecteurs soient porteurs de deutérium (Larsen, 1993 [6] ; Russel, 1996 [7]). Une autre application de la spectroscopie IR est le suivi de réaction directement sur une bille de résine[8],[9],[10]. Cette technique, bien que destructrice pour le support, permet de suivre qualitativement une transformation chimique. Cette technique comme la spectroscopie de masse nexige que très peu de matériel (une seule bille doit être aplati avant lacquisition afin dobtenir de bons spectres) pour une analyse, ce qui est très avantageux dans le cas du suivi de longues synthèses.
Une autre technique utilisée pour lanalyse structurale de composés ancrés sur support solide est la RMN du 13C. Cette technique ne nécessite aucune modification de léchantillon, ni de conditions particulières pour lanalyse. La seule préparation est le gonflement de léchantillon dans un solvant deutéré, ou protoné.[11],[12] Cette technique permet de déterminer le degré de fonctionnalisation des résines et permet également de suivre des synthèse supportées. La limite de cette technique est sa sensibilité. En effet, dû au faible rapport gyromagnétique du 13C (faible sensibilité par rapport au proton), et de sa faible abondance naturelle (1,11%), les temps dacquisitions sont très longs, ce qui limite le suivi de réaction sur le support. De plus, les spectres obtenus présentent une largeur de ligne assez importante. Dautres noyaux plus sensibles tels que le 19F et le 31P ont aussi été observés. [13], [14] La vraie limite de cette technique est quelle ne permet pas dobserver le noyau du proton, omniprésent dans les molécules organiques et avec un comportement tout à fait favorable à la RMN.
Le proton nest pas observable dans ces conditions à cause de la nature hétérogène des résines utilisées pour la synthèse en phase solide. De manière générale toutes les résines utilisées pour la synthèse en phase solide sont organisées de la même façon. On peut décomposer le support solide en trois zones.
Le premier domaine est la matrice solide qui constitue le corps principal du support solide. Ce support est en général une résine, cest à dire une matrice réticulée, constituée principalement de polystyrène réticulé par du divinylbenzène comme dans les résines Merrifield et Wang (zone 1 figure 5a), mais on trouve aussi des résines basées sur du polyéthylène glycol (POEPOP) et ou du polyacrylamide (Pega, PEPSYN).

Le second domaine est considéré comme le bras espaceur. Ce bras espaceur est
chimiquement inerte et de longueur variable (zone 2 figure 5a). La longueur du
bras est un facteur déterminant pour la mobilité des molécules et en
conséquence, aura une grosse influence sur la qualité des spectres RMN. Enfin,
à lextrémité du bras espaceur, se trouve le groupe fonctionnel qui va servir
dancrage à la molécule synthétisée (zone 3 figure 5a). La nature du groupement
chimique qui servira dancrage détermine le mode de coupure de la molécule
synthétisée du support solide.
Figure 5a : Représentation schématique dune résine pour la synthèse en phase solide
Les différents domaines qui constituent les résines
utilisées en synthèse organique en phase solide, en font un système hétérogène
avec des caractéristiques propres qui contribuent aux difficultés rencontrées
pour lanalyse de ce type de support.
Récemment une nouvelle technique RMN non destructrice a été introduite pour lanalyse directe de composés ancrés sur la phase solide. Cette technique basée sur la rotation de léchantillon à langle magique est la RMN MAS (Magic Angle Spinning). La RMN MAS est dérivée de la RMN du solide[15],[16]. La RMN MAS est une méthode danalyse directe et sensible ne nécessitant aucun traitement particulier de léchantillon si ce nest le gonflement de la résine dans un solvant deutéré approprié. La rotation (plusieurs kHz) à langle magique de léchantillon permet lobservation directe des noyaux 1H, 13C, et récemment 15N, avec une résolution proche de la RMN liquide haute résolution. Notre laboratoire sefforce daméliorer cette technique depuis plusieurs années.
2 La RMN HRMAS une technique de choix pour la synthèse organique en phase solide
2.1 Définition de la RMN Haute Résolution à lAngle Magique
La majeure difficulté rencontrée lorsque lon veut étudier une molécule ancrée sur un support solide par RMN est limportance de la largeur des raies. Dans le cas dun échantillon purement solide, la largeur de raie peut dépasser 10 kHz. Cet élargissement des raies provient notamment de linteraction dipole-dipole (interaction dipolaire). Une autre contribution à lélargissement des raies provient des inhomogénéités de susceptibilité magnétique. Ces variations de susceptibilité proviennent de lhétérogénéité de léchantillon, et peuvent être décrites comme une interaction dipolaire entre le spin et un dipôle macroscopique.[17]
Dans le cas dun échantillon de résine gonflée, il existe un gradient de mobilité allant de mouvements lents pour le polymère, vers des mouvements plus rapides pour la molécule ancrée sur le « linker ». Même si la mobilité des molécules ancrées à lextrémité du linker se rapproche des comportements rencontrés en solution, la largeur de raie reste importante et ne permet pas lobtention de spectres exploitables. De plus, si lon ajoute à cela une inhomogénéité importante de susceptibilité magnétique due à limportante hétérogénéité de léchantillon cette inhomogénéité contribue aussi à lélargissement. En effet lorsque la résine est gonflée, elle nest plus solide pur et comporte différentes zones, certaines contenant du solvant, dautres étant constituées de domaines non solubles à mobilité réduite, ou encore des domaines de grande mobilité. Toutes ces zones vont créer à lintérieur de léchantillon des grandes différences de susceptibilité magnétique, qui à leur tour vont contribuer à lélargissement de raies.
La RMN HRMAS appliquée à la synthèse en phase solide repose sur deux conditions. Contrairement à la RMN du solide, léchantillon doit être gonflé dans un solvant approprié. Le bon gonflement du polymère garantit une mobilité importante des molécules ancrées grâce à leur solvatation adéquate et réduit au moins partiellement les interactions dipolaires qui sont propres aux solides. Nous avons mentionné que dans le cas de la RMN gel, le simple gonflement de la résine permet lobtention de spectres 13C mais ne permet pas lobtention de spectres 1H. Du fait du rapport gyromagnétique élevé du proton, linteraction dipolaire et les effets de susceptibilité magnétique sont exacerbés et dans ce cas le gonflement de la résine nest pas suffisant pour moyenner ces interactions. La deuxième condition, est que lenregistrement des spectres doit être effectué en utilisant la technique de rotation de léchantillon à langle magique. Tandis que le gonflement de la résine permet une solvatation des molécules et leur confère ainsi une mobilité accrue par rapport à la résine sèche, la rotation rapide de léchantillon à langle magique permet de moyenner les différences de susceptibilité magnétique, ainsi que leffet dipolaire résiduel, ce qui permet dobtenir des spectres résolus en 1H, 13C pour la molécule ancrée sur le support.
En combinant le gonflement de la résine et le « magic angle spinning », le comportement des signaux RMN des molécules ancrées sur le support solide tend à se rapprocher des composés rencontrés en RMN liquide haute résolution (figure 5b). Toutes les séquences impulsionnelles utilisées sont celles empruntées à la RMN du liquide.
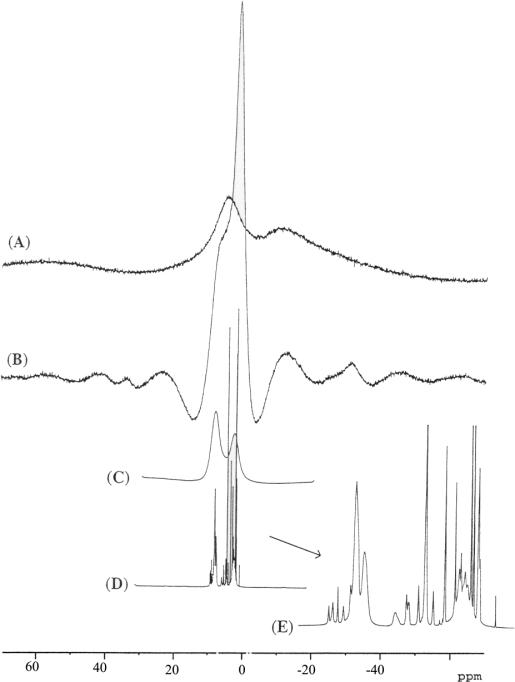
Figure5b : Spectres 1D 1H
de la peptidyl résine Wang-Ile-Val-Ser(OtBu)-Gly-Arg(Pmc)-Ala-H.
(A) Spectre statique de la peptidyl résine sèche. (B) spectre sous MAS (wr = 5kHz) de la peptidyl résine sèche. (C) Spectre statique de la peptidyl résine gonflée dans le DMF-d7. (D) et (E) spectre sous MAS (wr = 4kHz) de la peptidyl résine gonflée dans le DMF-d7.
2.1.1 Interaction dipolaire
Comme nous venons de le voir, linteraction dipolaire est en grande partie responsable de lélargissement important des signaux obtenus pour un spectre dune résine gonflée dans un solvant. Si lon représente graphiquement le champ dipolaire (figure 6) pour une paire isolée de spins, on constate quil existe une orientation pour laquelle le champ dipolaire, sans être nul, est perpendiculaire au champ magnétique statique B0.
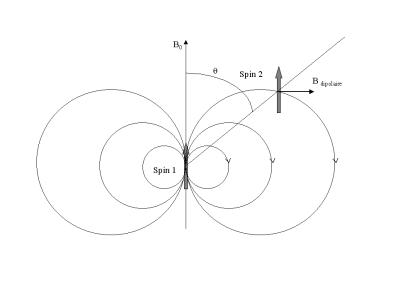
Figure 6 : Représentation graphique du champ dipolaire dun spin 1 orienté selon B0. Ce champ na plus de composante selon z quand q est de 54°,7 solution de 3 cos2(q)-1=0.
Linteraction dipolaire, qui est le produit scalaire du moment magnétique du spin avec le champ dipolaire est alors réduite à zéro. Lorientation pour laquelle linteraction dipolaire est réduite à zéro correspond à un angle de 54°,7 par rapport au champ statique B0, cet angle est appelé lAngle Magique. Le seul fait de basculer léchantillon à langle magique nest pas suffisant pour obtenir des spectres de qualité satisfaisante. Si lon considère un échantillon solide, il est évident que toutes les paires de spin ne peuvent pas toutes êtres orientées selon un axe orienté à 54,7° par rapport à B0. La rotation rapide de léchantillon selon un axe orienté à 54,7° par rapport à B0 va par contre imposer une orientation moyenne pour toutes les paires de spin qui coïncident avec laxe du cône de rotation. Ainsi lélargissement résiduel des raies (dû aux inhomogénéités du champ magnétique) est inférieur à 10 kHz, et est moyenné par la rotation rapide de léchantillon à une vitesse supérieure à linteraction que lon veut moyenner. Une rotation supérieure à 1kHz permet dobtenir des signaux suffisamment fins pour les molécules ancrées sur le support alors que les raies dues au polymère conservent une largeur importante de part leur mobilité restreinte. Cette différence de mobilité entre les signaux du polymère de la molécule ancrée sur le support et le solvant, peut être mise à profit lors des expériences HRMAS.
2.1.2 RMN HRMAS et le filtre de diffusion
La séquence dimpulsion du filtre de diffusion est une séquence dimpulsion permettant de saffranchir des signaux des molécules pouvant diffuser librement dans la matrice solide. Les molécules pouvant diffuser librement sont les molécules de solvants ou encore les excès de réactifs dans le cas de transformations incomplètes. Le séchage des résines pour le regonflement dans un solvant deutéré est une opération qui nécessite beaucoup de temps et il est parfois utile de pouvoir caractériser une molécule rapidement comme dans le cas dintermédiaires de synthèses sensibles, tels que les énolates.[18]
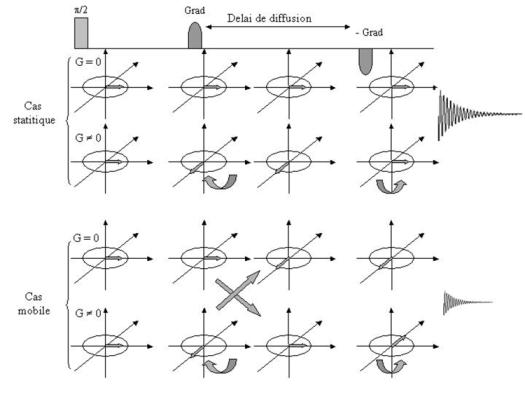
Figure 7 : Séquence dimpulsion simplifiée du filtre diffusion et son influence sur le vecteur aimantation.
Comme nous lavons vu précédemment, dans les échantillons de résines solvatées, il existe un gradient de mobilité entre les molécules ancrées sur la résine et les molécules libres en solutions. Dans certains cas ce gradient de mobilité peut être mis à profit. En effet les molécules greffées sur le support solide, bien que possédant une mobilité importante due à la libre rotation autour des liaisons, ont perdu la faculté de diffuser librement (mobilité translationnelle perdue de part lancrage sur la matrice solide) à lintérieur de léchantillon. Les molécules constituant le solvant, peuvent par contre diffuser librement dans tout léchantillon. Cette différence de mobilité translationnelle a été mise à profit au laboratoire pour supprimer les signaux dus au solvant[19]. La séquence dimpulsion du filtre diffusion repose sur lutilisation de gradients qui sont utilisés pour défocaliser laimantation (Figure 7).
Dans la figure précédente, on constate que pour les molécules ancrées (cas statique figure 7), lutilisation des gradients ne conduit pas à une diminution du signal car lors de lapplication du deuxième gradient, la molécule étant toujours à la même place, laimantation est re-focalisée. Par contre pour les molécules à forte mobilité, (cas mobile figure 7), lapplication du deuxième gradient ne re-focalise pas laimantation car pendant le délai de diffusion, les molécules se sont déplacées et ne ressentent donc pas le deuxième gradient avec la même intensité que le premier. Laimantation nétant pas re-focalisée, cela conduit à une perte de signal. Lorsque nous utilisons le filtre de diffusion au laboratoire, nous utilisons une puissance de gradient de 35 G/cm pendant une durée de 5 ms et un délai de diffusion de 30 ms. 35 G/cm représente 150000 Hz/cm. Lévolution de laimantation dun spin2 sur une molécule « libre » relatif à celle de dun spin1 au centre de léchantillon, sera différente par Dw.Dt ou Dw=2p.15000Hz. Le vecteur 2 tourne de 75 tours dans le référentiel ou le vecteur 1 est statique. Inversement nous pouvons calculer la distance par rapport au centre de léchantillon qui provoquera une simple inversion (évolution de laimantation p) de laimantation, le calcul nous donne une distance de 6µm. Si le délai de diffusion permet aux molécules mobiles de se déplacer sur une distance de 6µm au moins, lorientation de leur aimantation ne sera plus corrélée avec celle de la molécule au centre et nous perdrons du signal. Le coefficient de diffusion de leau liquide est de 2e-9 m2/s. Pendant les 30ms du délai de diffusion, une molécule deau se déplace de10µm dans le sens de laxe du gradient. Ceci signifie que durant le délai de 30ms, les molécules mobiles auront suffisamment de temps pour changer de place et ne plus voir leur aimantation refocalisée. Le filtre diffusion en RMN HRMAS permet donc de saffranchir des signaux dus aux solvants, ce qui se révélera très utile pour les études présentées dans la suite de ce chapitre.
La technique RMN HRMAS permet donc davoir un accès direct à lidentification complète des molécules attachées au support solide en cours de synthèse. Il est aussi possible de déterminer les problèmes structuraux qui peuvent être à lorigine de couplages difficiles[20]. Enfin, une analyse conformationnelle de la molécule basée sur lanalyse des contacts NOE (Würtrich, 1986)[21] et sur la déviation des déplacements chimiques 1H, 13C et 15N ( Wishart, 1992, 1994, 1995, Fruchart 2000)[22],[23],[24],[25] peut également être effectuée. Létude conformationnelle sur support solide peut avoir un avantage considérable sur la RMN haute résolution liquide pour les séquences peptidiques ayant une forte tendance à lagrégation.
2.2 Applications de la RMN HRMAS à la synthèse organique en phase solide
Récemment la technique RMN HRMAS combinée au gonflement de la matrice solide, sest révélée être la méthode danalyse de choix pour caractériser les composés organiques sur support solide. Au laboratoire, nous avons déjà montré que la technique permettait de suivre une transformation chimique,[26] lutilisation du filtre de diffusion a permis deffectuer le suivi en solvant protoné,17 et récemment nous avons montré que cette technique permettait la quantification des espèces impliquées dans une transformation chimique25. Contrairement à la spectroscopie de Masse ou à LIR, la RMN HRMAS ne permet pas de travailler à haut débit notamment à cause du temps de préparation de léchantillon. Comme nous lavons vu dans le chapitre dintroduction, la synthèse en phase solide est un outil intéressant pour la synthèse de molécules complexes mais aussi pour lobtention des banques de molécules de façon combinatoire. Comme nous voulons mettre au point une technique danalyse sur support solide, il faut répondre à deux questions : comment suivre une réaction de façon quantitative et comment détecter rapidement les impuretés éventuelles ? Les récents développements de la RMN HRMAS au laboratoire ont permis de répondre à la première question concernant le suivi quantitatif dune réaction, mais le problème de la détection rapide dimpuretés reste entier. Dans les paragraphes suivants nous verrons comment la RMN HRMAS peut apporter une solution à ce problème et nous nous intéresserons également à lanalyse de supports macroscopiques qui connaissent une utilisation croissante.
2.2.1 Suivi dune condensation de Wittig-Horner Emmons sur un support macroscopique.
Notre groupe a déjà appliqué la RMN HRMAS pour le suivi de réaction sur les résines classiquement utilisées en SOPS. [27] La manipulation de ces résines pour le suivi de réaction nest pas toujours évidente, en particulier dans le cas de réactions rapides. Pour suivre une réaction sur les résines, il faut prélever des aliquots à intervalles réguliers pour les introduire dans le rotor HRMAS afin denregistrer les spectres. Le remplissage du rotor nest pas toujours aisé et lacquisition des spectres nécessite du temps. Pendant lacquisition des spectres sur léchantillon prélevé, la réaction se poursuit. Les prélèvements successifs de quelques milligrammes de résine peuvent déséquilibrer la réaction, de plus les aliquots ne représentent pas la totalité du milieu réactionnel et on peut faire face à des problèmes de distribution statistique.
Comme nous lavons vu dans le chapitre dintroduction, le développement de la chimie combinatoire a contribué au développement de nouveaux supports solides, tels que les PINS ou les lanternes développées par Mimotopes. Ces nouveaux supports sont très utilisés en particulier dans le cas de la stratégie « one bead one compound ». De plus ces nouveaux supports ont trouvé un intérêt particulier dans le cadre des synthèses automatisées. Compte-tenu des difficultés déjà importantes de suivi de réaction sur les supports solides classiquement utilisés en phase solide, il est important de voir si ces nouveaux supports permettent une analyse directe des molécules ancrées sur leur surface. Lanalyse directe de molécules sur ce type de support a déjà été réalisée par dautres techniques telles que la spectrométrie de masse4 ou encore lIR[28]. Shapiro et col[29]ont montré quil était possible dobtenir des spectres 1D et 2D par RMN HRMAS complètement exploitables sur ce type de support mais ceci à laide dune sonde 7 mm et de plus après échange du solvant avec un solvant deutéré. Récemment Gerritz et al[30] ont étudié des copeaux de lanternes dans une sonde 4 mm. Bien que la sonde 4 mm soit un équipement plus conventionnel que la sonde 7 mm, le fait de raser les lanternes est une méthode destructrice et se rapproche du cas du suivi de réactions sur les résines conventionnelles.
Il est vrai que les dimensions actuelles des supports macroscopiques tels que les lanternes ne sont pas compatibles avec une étude directe de ce type de support dans un rotor 4 mm (figure 8).

Figure 8 : De gauche à droite : Capuchon de rotor 4mm, rotor HRMAS 4 mm, lanterne synphase ( 35 mmoles), microbeads (1400 nmoles).
Compte-tenu de ce problème de dimension, nous avons coupé une lanterne en quatre dans le sens de la hauteur. Bien que cette coupure du support ne soit pas satisfaisante, elle nous permet de conserver la nature macroscopique du support. Le quart de lanterne ainsi obtenu a été directement utilisé pour effectuer le suivi RMN HRMAS de la réaction de Wittig-Horner Emmons (Schéma 1), afin de comparer nos résultats avec ceux obtenus lors du suivi de la même réaction sur une résine standard.
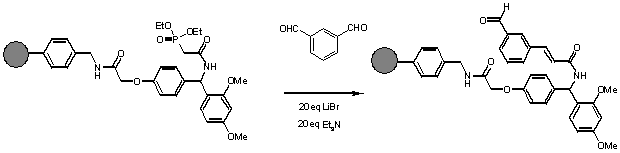
Schéma 1: Condensation de Wittig-Horner Emmons réalisé sur une lanterne SP-PS-D-RAM (fonctionnalisée par une linker Rink 35 mmoles).
Comme le montre le schéma réactionnel précédent, le térephtalaldéhyde a été ajouté sur le phosphodiester ancré sur la résine et nous avons procédé au suivi RMN afin de mettre en évidence lapparition du signal de laldéhyde qui nest pas impliqué dans la condensation.
Dans un premier temps, nous avons enregistré des spectres 1D dans le DMF deutéré afin de déterminer les conditions optimum pour effectuer le suivi de la réaction. Les spectres obtenus donnent des résultats satisfaisants mais afin doptimiser les paramètres du filtre diffusion, nous avons aussi enregistré des spectres du quart de lanternes dans les conditions du filtre diffusion. En accord avec les résultats précédemment décrits25, un délai de diffusion de 28 ms ainsi quune puissance de gradients de 35 G/cm, donnent des résultats satisfaisants. Non seulement le filtre de diffusion permet une suppression efficace des signaux du solvant, mais il permet aussi datténuer fortement lintensité des signaux de la matrice polymère. Cette intensité habituellement atténuée par lutilisation de la séquence CPMG[31],[32], est ici largement atténuée par la relaxation T2, lors de lapplication des gradients (figure 9). La séquence CPMG consiste en une suite dimpulsions 180° qui ont pour but de refocaliser laimantation. Durant lapplication de ces impulsions, laimantation se trouve dans le plan xy et en conséquence, on aura une perte daimantation par relaxation T2 pour les molécules à faibles mobilités. Dans le cas du filtre diffusion, la même perte daimantation par relaxation T2 se produit lors de lapplication des gradients. Après la phase doptimisation en solvant deutéré, nous avons de nouveau analysé léchantillon en solvant protoné. Là encore, le filtre diffusion permet de supprimer efficacement les signaux du solvant mais aussi atténue lintensité de la ligne due à la matrice polymère.
Pour effectuer le suivi de la réaction, le quart de lanterne a été introduit dans une solution contenant laldéhyde, puis la base et le catalyseur ont été rajoutés. Avant lintroduction de la lanterne dans le spectromètre le quart de lanterne a été abondamment lavé avec du DMF afin de stopper la réaction. Un lavage abondant de la lanterne est très important pour éliminer totalement laldéhyde nayant pas réagi. En effet, lors du premier essai, le spectre que nous avons obtenu présentait un signal daldéhyde double. Un nouveau lavage de lanterne nous a permis déliminer le signal provenant de la fraction soluble du térephtalaldéhyde (figure 10).
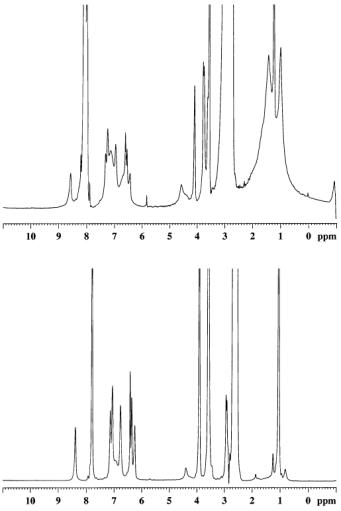
Figure 9 : Comparaison des spectres 1D dun quart de lanterne SP-PS-D-RAM, avant réaction. Lacquisition des spectres a été effectuée sur un spectromètre Bruker DMX 600. En haut le spectre présente des signaux intenses à 1,5 ppm provenant de la matrice polymère. Ces signaux sont largement atténués dans le cas du filtre diffusion (bas).
Comme la lanterne représente la totalité du milieu réactionnel le fait de la laver efficacement conduit à un arrêt de la réaction, ce qui permet de supprimer les contraintes de temps pour la préparation de léchantillon RMN. Dans lintroduction de ce chapitre nous avons discuté de différentes techniques danalyses et nous avons notamment abordé la notion de destructeur. Le terme de destructeur peut être considéré de deux façons : Soit la technique ne nécessite que très peu de matériel mais le support analysé est perdu pour la suite de la synthèse, soit lanalyse nécessite quelques milligrammes de résines mais le support est réutilisable. Le problème rencontré avec les résines est que les transferts du milieu réactionnel vers le rotor HRMAS ainsi que les transferts inverses, conduisent inévitablement à des pertes. Lors dune synthèse multi-étapes longue, les transferts engendrent trop de pertes ce qui nest pas satisfaisant. La nature macroscopique des lanternes ne conduit à aucune perte de matériel ce qui est très avantageux au regard des résines conventionnelles. Afin dobtenir un bon rapport signal sur bruit en vue dune intégration future, nous avons enregistré chaque spectre avec 256 scans. Après chaque spectre, le quart de lanterne était de nouveau introduit dans le milieu réactionnel. Lavancement de la réaction a été mis en évidence en mesurant lintégrale du pic de laldéhyde au cours du temps. Pour lintégration du pic de laldéhyde, nous navons pas besoin de références externes contrairement à létude sur la résine car dans le cas présent, la totalité de léchantillon se trouve dans le rotor (la quantité de produit observée reste constante) (figure 11). La courbe obtenue nest pas quantitative mais révèle seulement de lavancement relatif de la réaction. Un avancement quantitatif nécessite comme dans le cas de létude sur la résine, lemploi dune référence interne qui sert à étalonner la réaction.
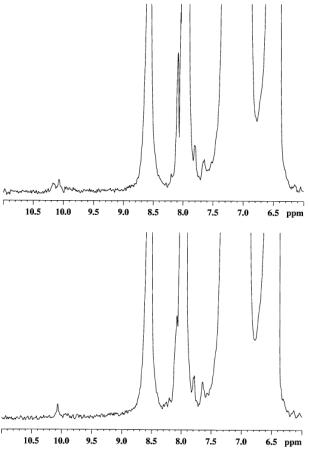
Figure 10 : Spectres 1D avec filtre diffusion, en haut lanterne après un simple lavage présentant un signal daldéhyde double dû à la fraction daldéhyde nayant pas réagi. Le spectre du bas ne présente plus cette résonance après un triple lavage de la lanterne par du DMF.
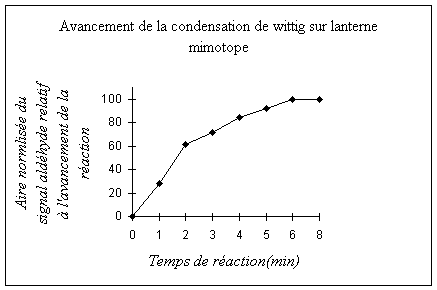
Figure 11 : Avancement de la réaction provenant de lintégrale du signal de laldéhyde
La forme de la courbe obtenue est en bon accord avec celle obtenue sur la résine, par contre dans le cas de la lanterne, la cinétique de réaction est plus rapide. En effet dans le cas de la résine la réaction était complète après 7h, dans notre cas la réaction est complète après 10 min. Cette différence concernant la cinétique de réaction peut trouver une explication dans la nature du support utilisé. En effet, la résine utilisée dans létude précédente est basée sur une matrice polystyrène qui par sa réticulation peut limiter la bonne diffusion des réactifs à lintérieur de la résine, alors que les lanternes sont normalement fonctionnalisées en surface. Une fonctionalisation uniquement basée sur une charge de surface ne semble pourtant pas être compatible avec une charge aussi élevée. Une étude de la structure des lanternes a été effectuée par spectroscopie RAMAN. Cette étude montre que la couche fonctionnelle a une épaisseur de 50 mm. Cette épaisseur de 50 mm correspond au diamètre dune bille dans les résines conventionnelles. Le polymère constituant la couche fonctionnelle est un polymère qui nest peu ou pas réticulé. Cette absence de réticulation permet une meilleure diffusion des réactifs dans la matrice polymère, ce qui peut expliquer la cinétique rapide constatée sur les lanternes par rapport aux résines conventionnelles qui elles sont réticulées.
Dans la première partie de notre étude nous avons mis en évidence lapparition dun signal daldéhyde. Cette résonance, qui a un déplacement chimique très caractéristique, permet donc une identification plus aisée. Comme toutes les synthèses organiques en phase solide ne sont pas toujours basées sur des fonctions ayant un déplacement chimique aussi caractéristique, il est important de déterminer si la spectroscopie à deux dimensions est possible sur ce type de support macroscopique. Afin didentifier toutes les résonances présentes sur la lanterne, nous avons enregistré un spectre TOCSY et un HSQC sur le quart de lanterne après réaction (Figure 12). Les spectres obtenus montrent que lidentification de molécules complexes peut parfaitement être effectuée sur ce type de support montrant une nouvelle fois limportance de lapport de la RMN HRMAS pour la chimie organique en phase solide.

Figure 12 : Spectres 2D sur léchantillon après condensation de Wittig-Horner. Le spectre TOCSY a été enregistré en DMF protoné. Le spectre HSQC 1H-13C a été enregistré en DMF-d7. Le pic de corrélation de laldéhyde est visible dans la zone marquée par une astérisque.
2.2.2 Détection dimpuretés en chimie organique en phase solide
2.2.2.1 Détection dimpuretés sur un support macroscopique
Avec le développement de la chimie combinatoire, le problème de la détection dimpuretés est devenu un enjeu crucial. Lors de la mise au point dun type de réaction sur support solide, il est impossible de tester les conditions de la réaction pour toutes les molécules qui vont être utilisées. Loptimisation est réalisée seulement sur quelques composés, en considérant que les conditions applicables à ces composés seront applicables à toutes les molécules de la même famille. La détection précoce dimpuretés dans le cadre de ces réactions tests est donc un enjeu dimportance pour éviter par la suite la présence de molécules non identifiées, potentiellement actives.
Après avoir montré que le suivi dune réaction même rapide était possible sur un support macroscopique, nous nous sommes donc intéressés à la détection dimpuretés sur ce type de support. Pour ce faire, nous avons donc couplé simultanément deux types daldéhydes sur la même lanterne. Afin déviter tous les problèmes liés à une différence de réactivité entre les deux espèces, nous avons couplé 1 équivalent dun mélange contenant 80% de 3-cyclohexen-1-aldéhyde et 20% de térephtalaldéhyde, pendant 10 min, cette durée correspondant au temps de réaction déterminé précédemment. Pour assurer un couplage total, 1 équivalent du mélange a de nouveau été engagé pour un autre couplage. Le spectre 1D permet la différenciation des deux espèces, de plus lintégration des différents pics conduit à une valeur de 81 % pour le produit de à la condensation du 3-cyclohexen-1-aldéhyde et 19 % pour le produit de condensation du térephtalaldéhyde (figure 13).

Figure 13 : Spectres 1D (temps de relaxation 3s) du quart de lanterne après couplage simultané des deux espèces. Lintégrale entre 5 et 6 ppm représente lespèce majoritaire, alors que le signal daldéhyde à 10 ppm représente lespèce minoritaire
Ces valeurs sont en bon accord avec le pourcentage théorique initialement introduit, montrant que la RMN HRMAS est une technique très intéressante pour lanalyse de réaction sur des supports macroscopiques.
2.2.2.2 Détection quantitative dimpuretés sur résine
Dans le cadre dune collaboration avec les Profs. Aubagnac et Martinez du LAPP de Montpellier (Laboratoire des Aminoacides, Peptides et Protéines), nous avons voulu tester les capacités de la RMN HRMAS pour la détection dimpuretés sur les résines couramment utilisées en synthèse organique en phase solide. En utilisant des quantités différentes de Glutamate et Pyroglutamate, Enjalbal et al 15 ont créé un système artificiel contenant un pourcentage contrôlé dimpureté. Létude de cette transformation nest pas anodine car ce type de modification de lacide glutamique peut se produire lors de la coupure du peptide en milieu acide. Ils ont ensuite procédé à lanalyse de leur système par spectroscopie de masse (SIMS) et ils ont comparé les résultats obtenus par cette technique avec les pourcentages déterminés par HPLC après séparation des molécules du support solide.5 Malgré la bonne corrélation entre les résultats de lanalyse SIMS et ceux obtenus par HPLC, confirmant la spectroscopie de masse comme une technique sensible pour la détection dimpuretés, la détection dimpuretés par la méthode SIMS pose certains problèmes notamment dus à des problèmes dionisation inhomogène ou encore dus à une distribution non homogène des molécules sur les différentes billes de résine, puisque lors de ces analyses, seules les molécules ancrées en surface sont accessibles.
Nous avons donc étudié le même système pour connaître les possibilités et limites de la RMN HRMAS vis à vis de la détection et quantification dimpuretés.
2.2.2.3 Identification des différentes espèces
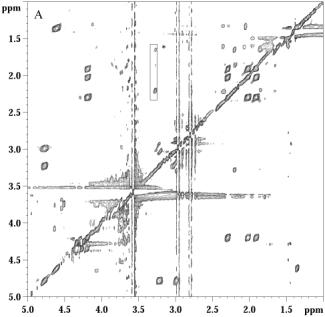
Avant de procéder à une étape de quantification, il est primordial didentifier les espèces en présence pour voir si le système est compatible avec une quantification ultérieure. Pour procéder à lidentification, nous avons enregistré les spectres TOCSY de trois échantillons, tout dabord les deux échantillons contenant 100% de chaque espèce ainsi que léchantillon contenant un mélange équimolaire de Glutamate et de Pyroglutamate (figure 14).
|
a |
|
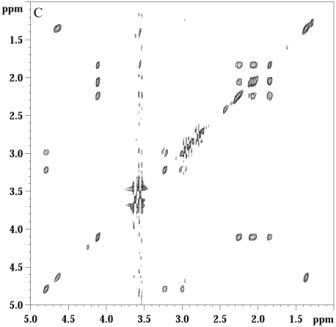
Figure 14 A) Spectre HRMAS TOCSY partie aliphatique sur 3 mg de léchantillon contenant 100% de Glutamate. B) Spectre HRMAS TOCSY partie aliphatique sur léchantillon, contenant un mélange équimolaire de Glu/Pyr. C) Spectre HRMAS TOCSY partie aliphatique sur 3mg de résine contenant 100% de Pyr.
Ces spectres nous ont permis didentifier toutes les résonances provenant de chaque acide aminé, de plus nous pouvons constater que les protons alpha du Glutamate et du Pyroglutamate ne résonnent pas à la même fréquence. La présence de résonances distinctes est un point crucial pour pouvoir procéder à une étape de quantification. En effet pour procéder à la quantification des différentes espèces, nous devons intégrer les signaux correspondants. Dans le cas où deux résonances sont très proches, lintégration des différents signaux peut être délicate et conduire à des résultats faussés. La nécessité dobtenir des signaux parfaitement séparés met en évidence le besoin de travailler avec des hauts champs. Si lon considère de nouveau les deux résonances voisines, elles ne seront pas suffisamment séparées sur un 300 MHz, mais elles le seront sur un 600 MHz.
2.2.2.4 Analyse quantitative des différentes résines
Pour procéder à la quantification des espèces, nous avons utilisé les spectres proton à une dimension des différents échantillons (figure 15). La proximité des protons CH du groupement protecteur Fmoc avec le proton alpha du Glutamate nétant pas favorable, nous avons utilisé le proton alpha du Pyroglutamate pour effectuer la quantification des différentes espèces. La quantification peut se réaliser très facilement par intégration du signal correspondant au proton a du Pyroglutamate par rapport à une référence. Il faut se remémorer que chaque échantillon contient un pourcentage variable de Glu et Pyr, mais contient par contre une quantité constante de Phe et de Ala (représentant dans tous les cas 100% des chaînes greffées sur la résine). Les protons alpha de lalanine ou de la phénylalanine peuvent alors servir de référence pour les intégrations. Nous avons donc utilisé ce principe pour notre étude et nous avons comparé nos résultats avec les résultats obtenus par spectroscopie de masse et aussi avec les valeurs de référence obtenues par HPLC après coupure (Tableau1).
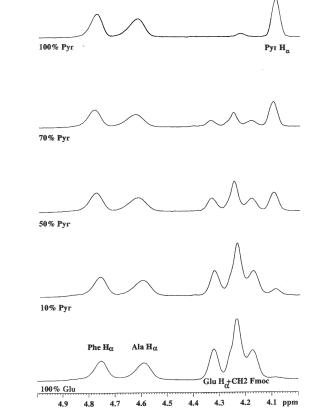
Figure 15 Spectre 1D des différentes résines gonflées dans le DMF-d7. Spectres enregistrés sur un spectromètre BRUKER DMX 600MHz avec 64 scans et un délai de 3s.
Après traitement des différents spectres à une dimension, nous avons obtenu des résultats proches des valeurs obtenues par HPLC après coupure, démontrant que la RMN HRMAS est un outil performant aussi bien pour lidentification que la quantification de molécules ancrées sur un support solide.
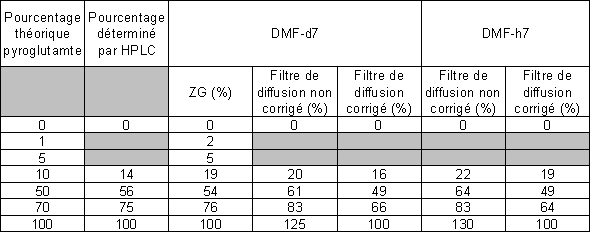
Tableau 1 : Tableau récapitulatif des pourcentages de pyroglutamate déterminés par les différentes techniques.
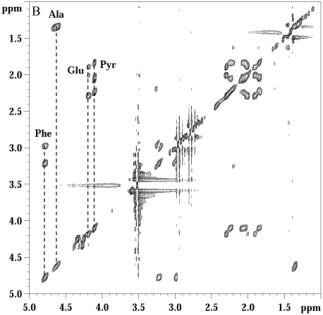
Après ces résultats concluants, nous avons voulu déterminer le seuil de sensibilité de notre technique. Pour ce faire, nous avons réalisé un échantillon contenant 1% de pyroglutamate. Après coupure, nous navons pas pu mettre en évidence la présence de pyroglutamate par HPLC. Par contre lacquisition dun spectre à 1D dans les mêmes conditions que précédemment, nous à conduit à une valeur de 2%. Pour déterminer les limites de la technique, nous avons effectué lacquisition dun spectre TOCSY (acquisition sur une nuit 72 scans) sur 10mg de la résine contenant 1% de pyroglutamate. A notre surprise, le spectre ne montrait pas seulement les corrélations dues au pyroglutamate, mais aussi un système de connectivité très proche de celui du glutamate (figure 16). Compte tenu du fait que lintensité de ce système de spin est de lordre de 1%, nous avons attribué ces résonances à la fraction de Glu se trouvant à la surface des billes de résine. De plus, les mêmes résonances mineures sont visibles pour léchantillon contenant une quantité équivalente de Glu et de Pyrglu , ainsi que pour les résonances des Ha de lalanine et de la phénylalanine. Il est couramment admis[33] que seulement 1% de la charge totale se trouve à la surface des résines ce qui est en bon accord avec la valeur que nous avons déterminée par intégration du pic de corrélation.
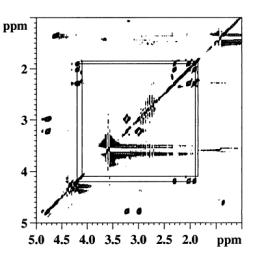
Figure 16 : Spectre TOCSY zone aliphatique sur 10 mg de résine contenant 1% de pyroglutamate.
La différence de déplacement chimique observée pour les molécules se trouvant en surface peut sexpliquer par une différence de susceptibilité magnétique. Les molécules se trouvant proches du cur de la résine se trouvent dans un environnement fortement aromatique dû à la présence du polymère, alors que les molécules de surfaces sont seulement en contact avec le solvant qui na pas un caractère aromatique. Afin de vérifier notre hypothèse, nous avons enregistré un spectre TOCSY sur la même résine mais en utilisant du benzène. Le spectre résultant ne présente pas ces résonances supplémentaires, ce qui confirmait lhypothèse de lobservation de molécules de surface. Dans cette expérience, le benzène a été utilisé pour mimer lenvironnement aromatique du cur de la résine et pour diminuer les inhomogénéités de champ magnétique. Un ré-examen des différents spectres 2D nous a permis de mettre en évidence une résonance inattendue dans le spectre de léchantillon contenant 100% de glutamate (figure 14A encadré). Nous avons attribué ce système de spin à une fraction de glutamate ayant été partiellement deprotégé. Pour contrôler notre hypothèse, une fraction de la résine a été déprotégée et le spectre obtenu présentait les mêmes résonances. Cette déprotection partielle du glutamate est due à un stockage prolongé de la résine dans du DMF. La mise en évidence de cette déprotection illustre bien lefficacité de la RMN HRMAS pour lidentification dimpuretés, mais aussi la sensibilité de la technique. On peut se demander si dans ce cas, les tests colorimétriques comme le test ninhydrine aurait pu mettre en évidence cette déprotection de la résine. Ces tests colorimétriques sont révélateurs de certaines fonctionnalités (ils ne sont donc pas universels) et peuvent dans certains cas conduire à des faux positifs ou à des faux négatifs. La sensibilité de ces tests vis à vis de la RMN HRMAS est à démontrer car ils conduisent à une réponse binaire alors que la RMN permet une identification complète des molécules ancrées. Toutefois les tests colorimétriques sont rapides et ne nécessitent que très peu de résines contrairement à la RMN.
Au début de notre étude, nous avons utilisé un solvant deutéré qui nécessite donc un séchage poussé de la résine pour ensuite la regonfler dans le solvant deutéré approprié avant dentreprendre les expériences RMN. Les phases de séchage et de regonflement de la résine sont des étapes qui nécessitent beaucoup de temps et qui ne sont pas compatibles en vue dune automatisation de ce type de technique. Nous avons donc utilisé le filtre de diffusion afin deffectuer notre étude dans un solvant protoné. Les études effectuées précédemment au laboratoire ont montré que le filtre de diffusion est une séquence moins sensible quune expérience « single pulse » notamment par une perte de magnétisation pendant le temps dapplication des gradients (laimantation se trouvant dans le plan xy), mais aussi par lévolution de la constante de couplage J et enfin due aux imperfections des impulsions. Il est à noter que ces facteurs ne sont pas constants pour toutes les molécules, comme par exemple la relaxation qui est plus longue pour le dernier résidu couplé[34]. Afin dévaluer linfluence de la séquence LED sur notre système, nous avons tout dabord utilisé cette séquence en solvant deutéré. Les résines contenant seulement un des deux composées nous ont permis de déterminer le facteur de correction à appliquer pour utiliser le filtre de diffusion dans le cas des échantillons se trouvant dans le solvant protoné. Ce facteur de correction a été déterminé par calcul du rapport entre lintégrale du Ha du pyroglutamate avec la moyenne des intégrales des protons alpha de lalanine et de la phénylalanine dans le même spectre de diffusion. Une fois ce rapport calculé, nous avons pu déterminer les différents pourcentages de la même manière que dans le cas précédent. Du fait de la moindre sensibilité du filtre de diffusion, nous navons pas pu déterminer les pourcentages des échantillons comptant respectivement 1% et 5% de pyroglutamate, dans les conditions que nous nous étions fixées (tableau1).
2.2.3 Limite de détection
Avec le développement de la chimie combinatoire, il est très important de disposer de méthodes de criblage de banques de molécules organiques. Les méthodes de criblage doivent être faciles à mettre en uvre, sensibles et rapides. De plus en plus les chimistes sorientent vers une bille un composé (« one bead one compound »), il faut donc disposer dune méthode danalyse possédant une sensibilité suffisante pour pouvoir observer le composé ancré sur une bille de résine.
Le rayon des billes constituant les résines couramment utilisées en synthèse organique en phase solide est très variable pour un même échantillon de résine et il est donc difficile destimer la charge dune bille de ces résines. La société LCC quant à elle fourni des résines dont le rayon des billes est hautement contrôlé et ce qui nous permet destimer la charge pour une bille de résine (figure 18).

Figure 18 : A gauche photo des billes dune résine de Merrifield. A droite LCC dynosphère
Si lon considère une résine de charge 0,4 mmol/g, on peut estimer que 1g de résine est constitué de 225 000 billes, ce qui conduit à une charge par bille de lordre de 2 nanomoles. Cette charge très faible ne nous a pas permis de mettre en évidence les signaux de la molécule ancrée sur ce type de support. Pour surmonter ce problème de charge insuffisante, nous avons utilisé des billes de résines dont la charge initiale à été augmentée par un processus de dendrimérisation. La synthèse de ces billes consiste à coupler sur les résines, des molécules polyfonctionnelles afin daugmenter la charge initiale de la résine. En fonction du degré de substitution des différentes billes, on dispose de billes de charges différentes (schéma 2). Ces billes de charge élevée sont des équivalents des lanternes dont nous avons discuté au début de ce chapitre, puisque lobjectif est dobtenir des objets aisément manipulables et possédant la charge la plus élevée possible. Les billes que nous avons utilisées ont été mises au point par le Dr Christophe Fromont au cours dun stage post-doctoral dans le laboratoire du Pr Marc Bradley (Université de Southampton). La taille des billes utilisées était denviron 500mm avec de grandes variations autour du rayon moyen. La charge des différentes billes variait de 50 nmol à 180 nmol par bille.
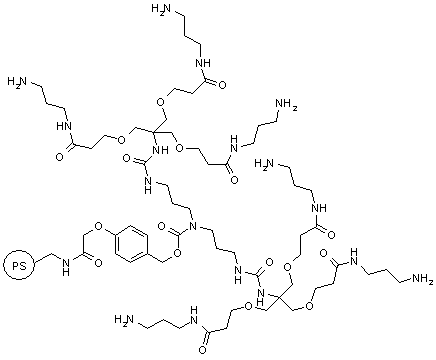
Schéma 2 : Formule développée plane dun dendrimère de 3éme génération de charge initiale 180nmol par bille.
Afin de développer une méthode de suivi de réaction nous nous sommes limités à 64 scans pour lenregistrement des spectres. Dans un premier temps nous avons couplé un acide aminé sur un lot de billes puis nous avons procédé à une condensation de Wittig sur un dialdéhyde. Nous avons choisi deffectuer une condensation de Wittig sur un dialdéhyde afin dobtenir une résonance parfaitement isolée et clairement identifiable. Lenregistrement des spectres avec 64 scans (sur 5 billes chargées à 180nmol) à mis en évidence la présence de laldéhyde et lenregistrement dun spectre avec filtre diffusion nous a permis de savoir avec certitude que laldéhyde était parfaitement ancré sur le support solide (figure 17).
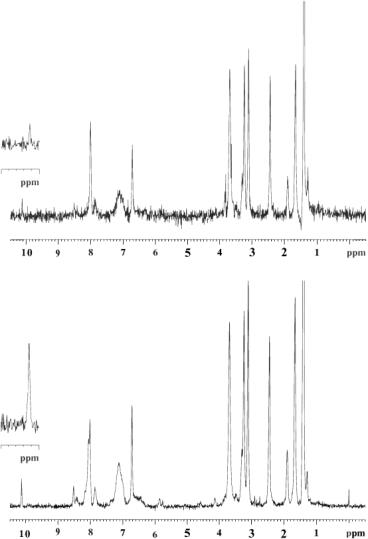
Figure 17 : Bas spectre enregistré sur 5 billes de dendrimère (180nmol par bille) après condensation de Wittig. Haut Spectre de diffusion du même lot de bille.
Afin de pousser encore plus loin notre technique, nous avons tenté le suivi de réaction sur une bille individuelle pour connaître le seuil de détection en terme de charge de la RMN HRMAS. Lenregistrement en 64 scans des spectres sur les billes de charges 180 et 100 nmol, permet dobtenir des spectres difficilement exploitables en utilisant une séquence dimpulsion classique, et lutilisation du filtre diffusion ne nous permet pas dobtenir des spectres de qualité suffisante pour procéder par la suite aux différents travaux dinterprétation. Les billes chargées à 50 nmol ne permettent pas lobtention de spectres exploitables que ce soit avec une séquence impulsionelle classique ou avec le filtre diffusion. Ce résultat est assez surprenant compte tenu des spectres obtenus par Duus et al 2000[35] sur des billes chargées à 0,9 nmol. En effet, en 512 scans ils obtiennent des spectres parfaitement exploitables avec des largeurs de lignes proches de la RMN haute résolution liquide. En dehors du matériel utilisé pour lacquisition des spectres qui ne peut expliquer à lui seul une telle différence dans les résultats obtenus La différence de qualité des spectres obtenus peut sexpliquer par la structure des résines elles-mêmes. En effet, de nombreuses études ont montré que la structure des résines et les solvants utilisés ont une grande influence sur la qualité des spectres. Une étude récente[36] a montré que les résines entièrement basées sur du polyéthyléneglycol présentent un meilleur gonflement que les résines basées sur du divinylbenzène. Le gonflement des résines étant étroitement lié à la mobilité des molécules ancrées sur le support solide, cela se traduit immédiatement sur la qualité du spectre en terme de largeur de lignes, mais le paramètre qui contribue le plus a lélargissement des lignes est sûrement la présence daromaticité dans la structure de la résine.[37] La présence des cycles aromatiques dans la structure de la résine est à lorigine dun champ magnétique local induit par le mouvement des électrons. Un proton situé au voisinage des ces cycles aromatiques va donc ressentir un champ magnétique différent suivant lorientation des cycles. La variation du champ magnétique local autour du proton va donc induire des déplacements chimiques différents qui conduisent à lélargissement des lignes. Dans leur étude Duus et al ont utilisé des billes de résine POEPOP1500. Ce type de résine, comme nous lavons déjà vu précédemment, est particulièrement favorable pour une étude RMN HRMAS, puisque ces résines sont totalement dépourvues de cycles aromatiques. Le relatif échec que nous avons rencontré pour déterminer la limite de détection en terme de charge de la RMN HRMAS, est dune part lié au type de support utilisé, mais aussi aux conditions dacquisition. En effet, nous nous sommes limités à 64 scans pour développer une méthode permettant le criblage à haut débit, alors que Duus et al, ont utilisé 8 fois plus de scans. Toutefois la différence de qualité entre les spectres que nous obtenus sur les dendrimères et la qualité des spectres obtenus sur des billes de POEPOP1500 nest pas uniquement liée au nombre de scans mais plus particulièrement au support utilisé.
3 Conclusion
Dans ce chapitre nous avons vu que la RMN HRMAS peut apporter une réponse intéressante aux problèmes rencontrés pendant la SOPS. Cette technique permet dune part détudier les nouveaux supports développés pour la synthèse organique en phase solide, et permet aussi de suivre des transformations rapides sur ce même de type de support, sans nécessité de séparer les molécules du support, mais aussi sans aucune modification de léchantillon. De plus, nous avons aussi montré que la RMN HRMAS était une technique sensible pour déterminer précisément le pourcentage dimpuretés formées au cours dune réaction chimique sur différents supports solides. Toutefois cette technique, malgré sa grande efficacité pour étudier des milieux hétérogènes, est limitée par sa sensibilité en terme de charge et la qualité des spectres obtenus est hautement liée au support utilisé.